科学家开发新方法描述基因表达动态过程
单细胞测序技术描绘出的高维基因表达图谱可以提供细胞状态的“快照”,如何从这些静态“快照”中准确地推演细胞的动态轨迹,是生物信息学中的一个重要问题。中国科学院过程工程所生物药制备与递送全国重点实验室联合上海交通大学开发了RNA速度建模新方法——TSvelo。这一方法不仅能描述未剪接和剪接RNA的动态变化,还能将转录调控信息进行联合建模,从而实现基因表达全过程动力学刻画。相关工作近期在eLife上发表。
近年来,研究人员利用人工智能技术开发出了一系列细胞轨迹模型,用于研究细胞如何在发育、分化或疾病状态下做出命运选择。其中,RNA速度模型通过对RNA剪接这一动态过程进行建模,推测细胞潜在时间和未来状态。然而,现有方法在构建模型时大多依赖于未剪接与剪接后RNA的丰度关系,不仅难以应对剪接信号稀疏、噪声干扰显著的技术难题,更缺乏对高维基因间复杂调控网络的系统性整合,限制了预测的准确性和可解释性。
针对这一挑战,研究团队基于神经常微分方程(Neural ODE)开发了TSvelo模型,系统处理转录调控、转录、剪接和降解等多重过程中的复杂连续变化。Neural ODE中刻画了大量具有明确生物意义的模型参数,包括转录率、剪接率、降解率及调控权重等,从而兼顾了模型的可解释性和预测精度。模型采用期望最大化算法的计算框架,通过迭代优化Neural ODE参数和细胞潜在时间,学习高维基因表达的时序演化模式与潜在调控关系,最终实现对细胞命运的精准预测和深入解析。
在实际应用中,TSvelo能够精确拟合单基因的表达动力学,并生成各个基因的转录率-未剪接-剪接三维相图,直观展示基因表达随时间的变化模式。结合RNA速度流向分析,TSvelo能够预测细胞未来状态,重建细胞迁移和分化趋势。此外,模型还具备多分支和初始点鉴定功能,可以成功区分不同谱系细胞的分化轨迹,并提示关键转录因子在不同分化路径上的动态调控作用。在多个单细胞数据集上的测试结果显示,TSvelo在预测结果的连贯性等方面优于目前主流方法,展示出TSvelo具有广泛的应用前景,可用于解析发育过程、细胞分化和转分化机制等基础研究。
过程工程所工程师李家琛为论文第一作者,过程工程所研究员袁野为通讯作者。
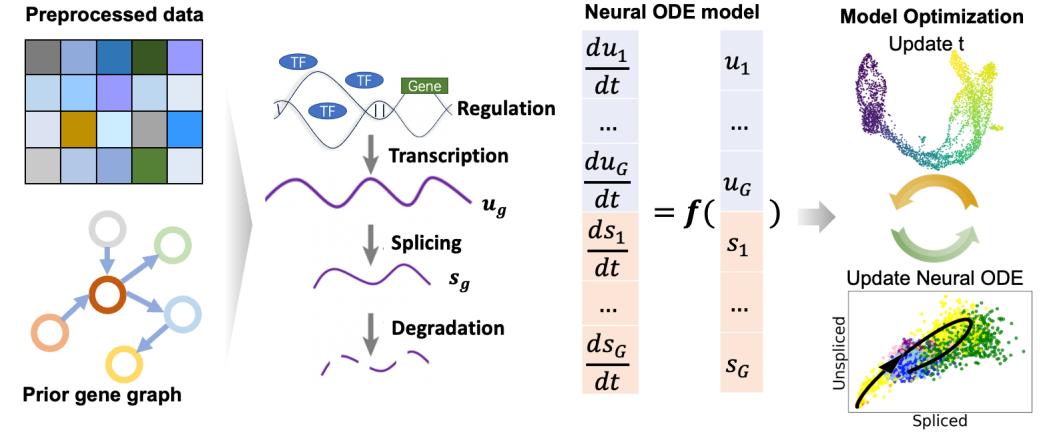
TSvelo模型流程示意图。研究团队供图
相关论文信息:https://doi.org/10.7554/eLife.108950.1



